南海包括大量从海底隆起的山脉,称为海山,其独特的海洋环境可以改变周围水域的水动力学,产生“海山效应”。海山区上升流可将富含营养盐的深层海水带入真光层,从而提高海山周围海域的初级生产力和生物多样性,为浮游生物创造了独特的栖息地。海山效应影响了海洋浮游植物的分布格局,并可能会进一步影响与浮游植物密切相关的异养微生物的群落结构,且微生物在海山区与陆架或沿海海域是否具有连通性仍有待深入研究。
研究团队通过参加2021年实验室与中山大学联合组织实施的 “南海科学考察计划”南海中部综合科考航次,从宪北海山区及近岸海域(东沙、西沙)收集了61个海水样品,采用扩增子测序技术进行了微生物多样性分析,结果显示,Alphaproteobacteria和Gammaproteobacteria在所有表层(5 m)海水样品中是最丰富的细菌类群。Cyanobacteria 主要分布在150 m以上的水层中。在宪北海山区随水深增加,Alphaproteobacteria和Cyanobacteria的相对丰度呈下降趋势,而Gammaproteobacteria、Thermoproteota、SAR324和Marinimicrobia(SAR406)的相对丰度则随水深呈增加趋势。
结合宏基因组测序、组装和分箱,该研究共获得了1887个非冗余基因组(MAGs),其中153个为符合MIMAG标准的高质量MAGs。物种注释结果显示,约240个MAGs为古菌基因组,1647个为细菌基因组。细菌基因组可以分为24个门,主要由Alphaproteobacteria(395)、Gammaproteobacteria(395)、Bacteroidota(157)、Actinomycetota(156)、Planctomycetota(127)、Verrucomicrobiota(93)、Chloroflexota(73)、Marinisomatota(67)和SAR324(65)等组成。
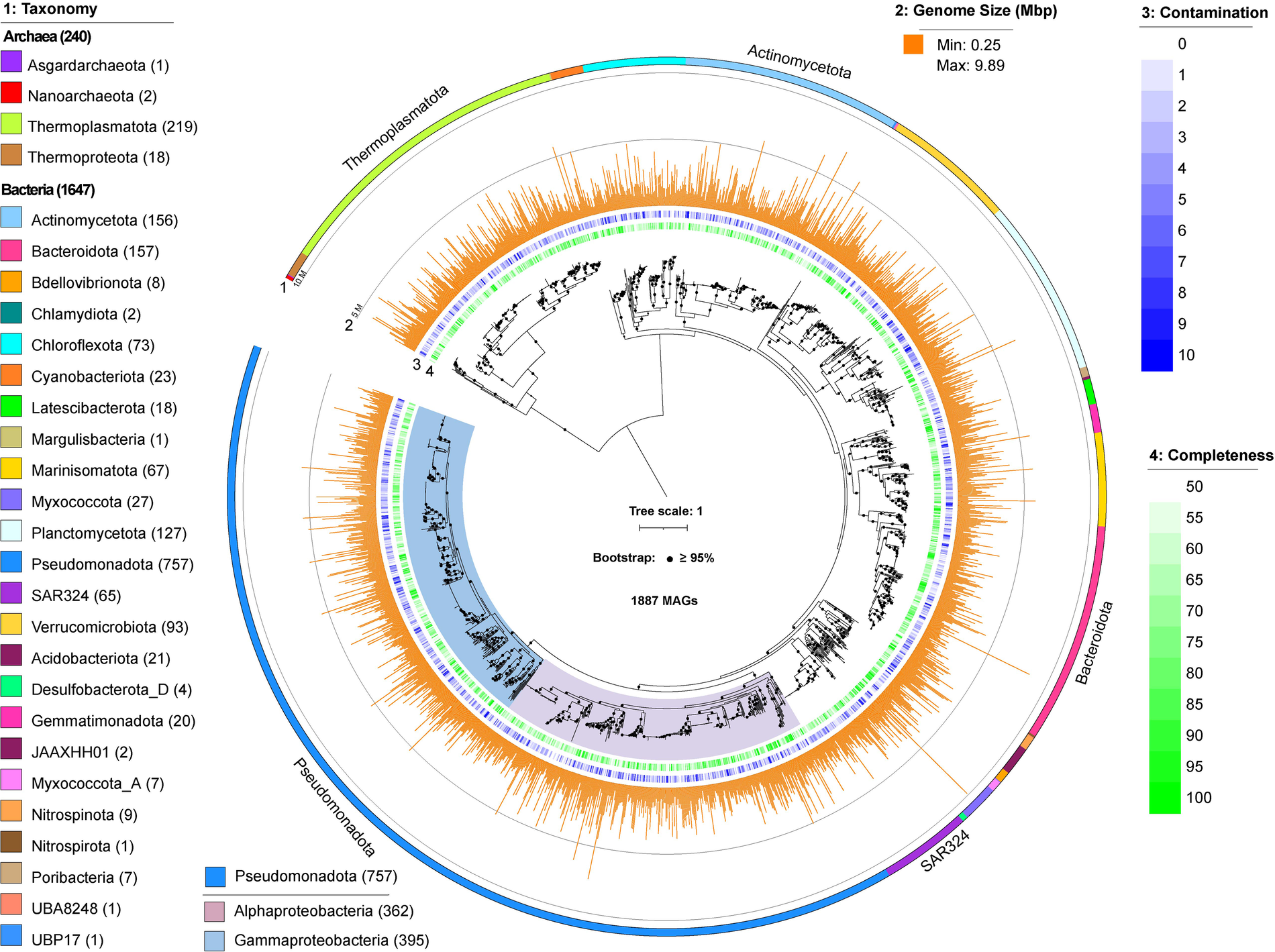
1887个MAGs构建的系统发育树
通过对1887个非冗余基因组与南海不同生境(冷泉、沉积物和河口)、OceanDNA和Tara Oceans等已发表的MAGs比较,研究发现366 MAGs未在其它参考基因组数据集中出现,说明本数据集构建的微生物基因组是对公众数据的重要补充。研究人员进一步基于contig构建了非冗余基因集,获得了10,551,413个代表基因,并使用KEGG Orthology (KO)对这些基因的功能进行了注释。该数据集提供的群落结构和基因组信息可用于分析南海微生物的分布和代谢功能。
该成果发表在Nature出版集团刊物Scientific Data(影响因子9.8),样品由 “南海科学考察计划”南海中部综合科考航次支持,深海生命与生态过程创新团队成员宁波大学姜海波教授和南方科技大学侯圣伟助理教授为文章的通讯作者,南方科技大学徐帅帅博士、宁波大学黄海龙助理研究员和南方科技大学陈松泽博士为共同第一作者,创新团队核心成员谢伟教授等对本研究具有重要贡献。
本研究得到了国家自然科学基金项目、南方海洋实验室自主科研项目等的支持。


